植物转录因子结合位点预测研究取得新突破
南湖新闻网讯(通讯员 辛西)近日,我校信息学院生物统计团队胡学海教授课题组研发出一款针对植物转录因子结合位点预测的工具及其docker镜像,相关研究成果发表在国际生物信息学领域学术期刊Bioinformatics上。
转录因子结合位点(TFBS)是顺式调控元件的基本组成部分,在基因表达的精确调控中起重要作用。TFBS核心基序内的非编码变异可能会显著改变其结合亲和力,这可能是解释遗传变异如何影响复杂性状的生物学机制。植物中转录因子结合位点实验数据的缺乏,以及植物TFs的独立进化特性都使得鉴定植物TFBS的计算方法落后于相关的人类研究。本研究首先使用深度卷积神经网络(DeepCNN)在基于可用的拟南芥Dap-seq数据集建立了265个拟南芥TFBS的预测模型,并且将其迁移用于预测其他植物的同源TF中。
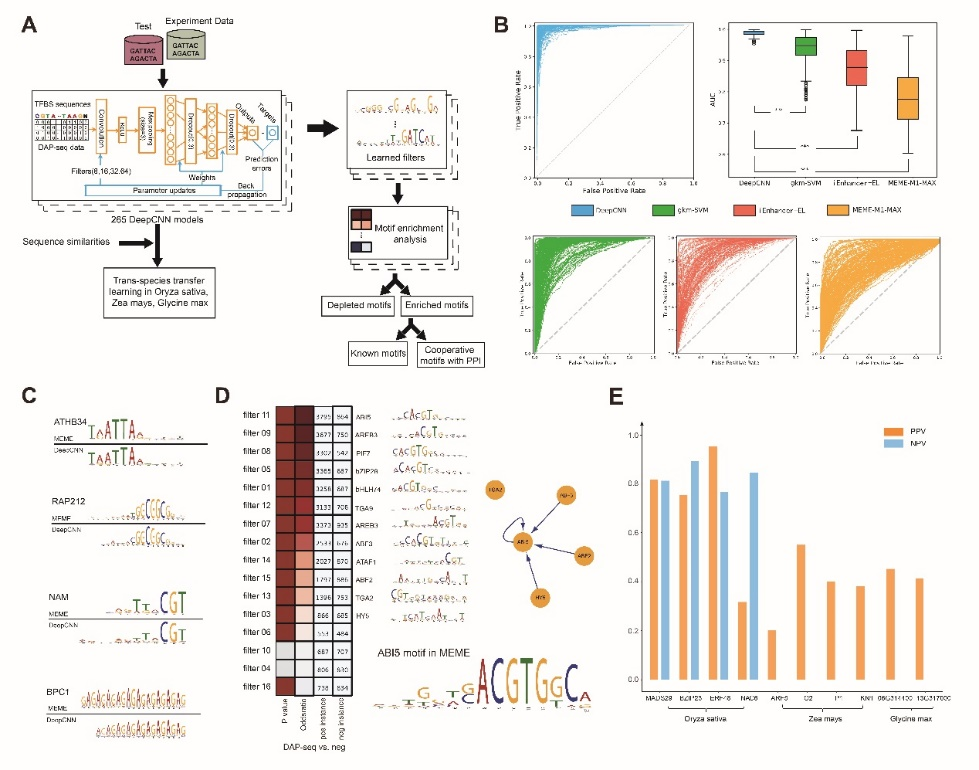
建模结果表明,DeepCNN在265个拟南芥数据集上都获得了很高的预测精确度(平均AUC达0.96),阐明了其在植物TFBS预测方面的可行性。通过进一步深入分析DeepCNN中卷积核的性质,作者提供了模型的生物学可解释性:DeepCNN不仅能学习到当前转录因子在序列当中的关键结合motif,而且能够学习到与该转录因子共同协作的转录因子的结合motif。
最后当使用迁移学习技术尝试从计算的途径解决目前植物TFBS研究问题的困难时,作者发现在不同的植物种类中,迁移学习的表现具有很大的不同。在水稻的十个TF中的三个都取得了比较好的预测效果,BZIP23 、ERF48和MADS29的 PPV(Positive predictive value)分别为0.752、0.951和0.816。而当迁移到玉米和大豆中时,预测效果均不甚理想。这表明迁移学习在植物的跨物种转录因子结合位点预测问题上具有一定的可行性,但是未来我们仍需设计更加有效的迁移学习策略。
为了提供更方便、更优质的生物信息学服务,课题组为此具有高精确率辨别转录因子结合位点的深度卷积神经网络模型搭建了docker镜像,通过下载该镜像并在本地配置可以实现离线预测植物转录因子结合位点的预测功能(https://github.com/liulifenyf/TSPTFBS)。
【英文摘要】
Motivation: Both the lack or limitation of experimental data of transcription factor binding sites (TFBS) in plants and the independent evolutions of plant TFs make computational approaches for identifying plant TFBSs lagging behind the relevant human researches. Observing that TFs are highly conserved among plant species, here we first employ the deep convolutional neural network (DeepCNN) to build 265 Arabidopsis TFBS prediction models based on available DAP-seq (DNA affinity purification sequencing) datasets, and then transfer them into homologous TFs in other plants.
Results: DeepCNN not only achieves greater successes on Arabidopsis TFBS predictions when compared with gkm-SVM and MEME, but also has learned its known motif for most Arabidopsis TFs as well as cooperative TF motifs with PPI (protein-protein-interaction) evidences as its biological interpretability. Under the idea of transfer learning, trans-species prediction performances on ten TFs of other three plants of Oryza sativa, Zea mays and Glycine max demonstrate the feasibility of current strategy.
Availability and implementation: The trained 265 Arabidopsis TFBS prediction models were packaged in a Docker image named TSPTFBS, which is freely available on DockerHub at https://hub.docker.com/r/vanadiummm/tsptfbs. Source code and documentation are available on GitHub at: https://github.com/liulifenyf/TSPTFBS.
Contact: huxuehai@mail.hzau.edu.cn
原文链接:https://academic.oup.com/bioinformatics/article/37/2/260/6069568
审核:胡学海
未经允许不得转载:大学门户 » 植物转录因子结合位点预测研究取得新突破
相关推荐
- 图书馆毕业季主题书展开展
- 【战“疫”故事】文心抗疫:景园2019级举行篆刻印稿特别设计
- 校党委理论学习中心组开展“学史增信”专题学习研讨
- 翻转课堂翻新样,最美声音进课堂
- 【战“疫”快报】爱心农企捐赠助力校园抗疫
- 华中农业大学在动物源性成分检测方法上取得新进展
- 华中农大为延期报到的41名新生举行开学典礼
- 器乐技艺普及班:学长为师 共同学艺
- 李召虎参加大学创新创业教育线上交流会并作专题报告
- 宋雨:勤学党史悟伟力,笃行求知以成人
- 学校管理与服务人员代表张智勇老师在2020年毕业典礼上的发言
- 我校在全球土壤地衣型真菌多样性与生态分布研究方面取得进展
- 校友企业增资“园艺新秀奖励基金”捐赠仪式在学校举行
- 湖北工业大学党委副书记蔡光兴来校交流
- 华中农业大学师生热议习近平总书记给复旦大学《共产党宣言》 展示馆党员志愿服务队全体同志回信精神
- 我校教师在2019新时代高校环境教学改革与创新研讨会上作报告
- 学校召开学院院长交流培训会
- 我校第五十九届田径运动会开幕
- 省科技厅长王炜来我校慰问新型肺炎应急科研攻关研究专家
- 我校4个2017年度湖北省技术创新专项重大项目通过结题验收
新闻公告
- 学校召开2022年网络安全和信息化领导小组会议 03-17
- 校学术委员会召开全体会议 03-17
- 学校部署2022年定点帮扶建始县工作 03-16
- 我校一名博士后入选第七届中国科协青年人才托举工程 03-16
- 异地同步 云端同窗 “慕课西行”课程开讲 03-16
- 学校一公共空间设计方案通过 03-16
- 机器学习和生物大数据交叉融合助力智能育种 03-15
- 校领导班子召开2022年第7次调度会 03-15
- 严建兵与信息学院同学们共同品味书香话成长 03-14
高考招生
- 2017年华中农业大学普通本科招生章程 08-05
- 2018年华中农业大学普通本科招生章程 08-05
- 华中农业大学2015年全日制普通本科招生章程 08-05
- 华中农业大学普通本科招生章程(2016年) 08-05
- 华中农业大学2013年全日制普通本科招生章程 08-05
- 华中农业大学2014年全日制普通本科招生章程 08-05
- 华中农业大学2012年全日制普通本科招生章程 08-05
- 华中农业大学2008年全日制普通本科招生章程 08-05
- 华中农业大学2009年全日制普通本科招生章程 08-05
- 华中农业大学2007年全日制普通本科招生章程 08-05
